【专题】第三代测序技术——单分子测序
前两年在木虫上发了一个二代测序的帖子,收到了不少虫友的回复,可见这个领域还是颇受关注的。由于个人原因,AFK了好久,哈哈......再回到论坛时,已经有了新的定位和方向。最近接触了很多单分子测序的信息,工作也与此相关,故再发一帖,与大家分享。
旧帖地址 【专题】第二代测序技术漫谈
--------------------------------------分割线---以下正文--------------------------------------
一、单分子测序简介
自从2006年第一台454 GS FLX测序平台上市以来,基于非Sanger测序原理的第二代高通量测序 (next-generation sequencing, NGS) 技术迅速成为了基因组学研究的重要工具,其中包括Illumina Solexa、ABI SOLiD、Roche 454以及Life Tech的半导体测序仪Ion Torrent PGM & Proton。这些平台原理各有不同,在通量、读长、准确度、速度和成本方面各具优势,均在基因组de novo,重测序、转录组、表观遗传学研究中发挥了重要作用,并逐渐应用于个性化医疗和遗传诊断等临床服务。近五年来,NGS的数据产出一直呈现出指数增长的趋势,平均每5个月数据量增加一倍。(数据来源:NCBI Sequence Read Archive)
另一类非Sanger原理的DNA测序技术在2008年成为现实,这类基于单个分子信号检测的DNA测序被称为单分子测序 (single molecule sequencing, SMS),或第三代测序 (third generation sequencing, TGS)。据预测,SMS将比NGS具有更快的速度和更低的成本,从而使研究人员能够实现目前无法进行的研究工作[1]。尽管从现在的进展来看,SMS还未能完全实现预期目标,但已经做出了许多重要的努力。这些新技术包括Helicos的tSMS,PacBio的SMRT,Oxford的Nanopore以及其它一些尚处于实验室阶段的技术,如电镜测序,蛋白质晶体管测序等等。
二、tSMS(ture single molecule sequencing)
Helicos Bioscience (MA, USA) 于2008年推出的HeliScope单分子测序平台被认为是第一个商品化的第三代测序仪。其测序原理tSMS是由斯坦福大学的S. R. Quake等科学家提出的。tSMS是一种利用光学信号进行DNA碱基识别的边合成边测序 (sequencing by synthesis, SBS) 技术,与二代测序中的Illumina Solexa测序有类似之处,但该技术无需对样本进行PCR扩增,简化了测序文库的构建过程,也避免了DNA扩增中出现的错误。HeliScope的文库制备相对简单,首先将待测DNA随机打断成约200bp大小的片段,然后在3’末端加上50bp带有荧光标记的poly(A) tail。文库退火形成单链,与芯片上固定的Oligo dT探针结合,利用poly(A)上的荧光标记进行精确定位。接下来依次加入4种Cy5荧光染料标记的单核苷酸,在DNA聚合酶的作用下与模板互补配对并延伸一个碱基,ICCD相机采集荧光信号。最后通过化学剪切去除荧光基团并清洗,进行下一轮反应。原理如图所示。
了解Illumina测序原理的同学对tSMS应该会有一种熟悉的感觉,二者原理和流程较为相似,所不同的在于tSMS采集的是一条DNA模板合成时所发出的荧光,而Illumina检测的信号来自于桥式PCR扩增得到的DNA(模板)cluster合成时发出的荧光。因此,tSMS技术能够实现单分子测序,主要依赖于高分辨率的ICCD相机,能够对单个分子产生的荧光信号进行识别。但是较弱的信号强度导致测序的读长较短,错误率偏高,尽管通过两次测序 (two-pass sequencing) 能够降低错误率,但同时也提高了测序成本和运行时间。HeliScope可同时运行两个芯片,平均读长约为35bp,一次运行的数据产量可达30Gb左右。该测序仪的售价和运行成本相对较高,一个人类基因组的测序成本约为5万美元。不过,该公司由于经营不善等原因(当然,也有测序仪本身性价比的原因),已经broke了。
参考文献:Harris TD, et al. Single-Molecule DNA Sequencing of a Viral Genome. Science 320: 106-109 (2008)(附原文)

图中显示的是HeliScope测序时的成像过程(右半部分),图中1-8均为6.6μm2大小同一位置的成像结果。画面1是利用poly(A)上的荧光标记进行定位,每一个光点均代表一条被固定在芯片上的文库模板。画面2-8显示了7轮反应的结果,依次加入G-C-A-G-T-C-A,根据图像可识别出位置1延伸的前4个碱基是5’-CGCA-3’[ Last edited by holyala on 2014-9-3 at 22:58 ]




 京公网安备 11010802022153号
京公网安备 11010802022153号
继续~~

三、SMRT(single molecule Real-Time)sequencing
单分子实时测序是Pacific Biosciences (CA, USA) 推出的一项专利技术。该方法采用四色荧光标记的dNTP和被称为零级波导 (zero-mode waveguides, ZMW) 的纳米结构对单个DNA分子进行测序。这种ZMW是直径50-100纳米,深度100nm的孔状纳米光电结构,通过微加工在二氧化硅基质的金属铝薄层上形成微阵列,光线进入ZMW后会呈指数级衰减,从而使得孔内仅有靠近基质的部分被照亮。Φ29 DNA聚合酶被固定在ZMW的底部,模板和引物结合之后被加到酶上,再加入四色荧光标记的dNTP (A555-dATP, A568-dTTP, A647-dGTP, A660-dCTP)。当DNA合成进行时,连接上的dNTP由于在ZMW底部停留的时间较长 (约200ms),其荧光信号能够与本底噪音区分开来,从而被识别。荧光基团被连接在dNTP的磷酸基团上,因此在延伸下一个碱基时,上一个dNTP的荧光基团被切除,从而保证了检测的连续性,提高了检测速度。SMRT的测序原理如图所示。
SMRT的一大优势是超长的读长,PacBio RS II测序平台能够得到的最大读长为30Kb,平均读长约8.5Kb,是目前所有商品化测序仪中读长最长的。一个SMRT cell单次运行产生的数据量约为400Mb,样本制备时间需要8-10 hr,测序时间45-90 min,如果同时运行多个SMRT cell,则一天能够产生最多6.4Gb的数据。与tSMS类似,因为单分子的荧光信号较弱,SMRT的单碱基准确率仅有87.5%,但由于错误是随机产生的,通过多重测序和校正,在10×的条件下,准确率可提高到99.9%。
参考文献:Eid J, et al. Real-Time DNA Sequencing from Single Polymerase Molecules. Science 323: 133-138 (2009)
图2A显示的是ZMW的结构,激光进入ZMW后呈指数级衰减,仅能照亮靠近底部的约30nm区域,因此大部分游离的荧光标记dNTP不会被激发,只有结合到DNA聚合酶上的dNTP其荧光基团被激光照亮,激发荧光。图2B显示的是DNA合成过程中检测到的荧光信号及持续时间。结合到酶上的dNTP停留时间较长,信号呈脉冲式激发,因而能够与噪音区分。
顶一下,感谢分享!
今晚搞定~~
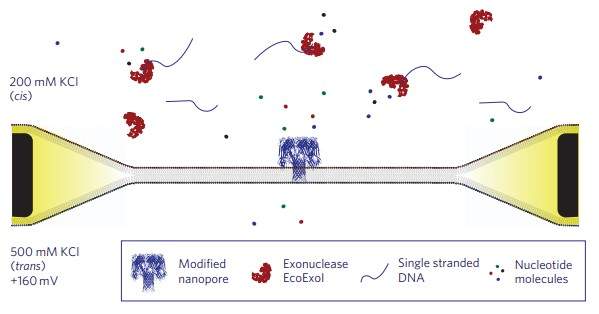
四、Nanopore
利用纳米孔进行核酸序列的识别在上世纪90年代已有报道。其基本原理类似于库尔特计数器 (Coulter Counter),当单链DNA或RNA分子通过纳米级的小孔时,由于碱基形状大小不同,引起孔内电阻变化,在小孔两端保持一个恒定的电压,则能够检测到通过小孔的电流变化情况,通过测到这些特征电流,就能够识别出通过小孔的DNA分子上的碱基排列。该方法具有检测速度快,成本低,准确率高等特点,但也面临DNA易位速率过快,电流变化幅度较小,制备纳米孔材料的稳定性等问题。
Oxford Nanopore Technologies (Oxford, UK) 应用这一原理开发了两种纳米孔测序技术:外切酶测序 (Exonuclease sequencing)和链测序 (Strand sequencing)。外切酶测序是将α-溶血素和环化糊精组成的纳米孔固定在脂质双分子膜上,两侧为浓度不同的KCl溶液,并加以160mV的电压。DNA单链在E. coli核酸外切酶I的作用下被依次剪切为单核苷酸,通过记录单核苷酸分子通过纳米孔时引起的电流变化进行DNA测序。链测序则是利用DNA解旋酶将DNA双链解旋为单链,并通过纳米孔,进行连续测序。目前Oxford Nanopore Technology推出的GridION和MinION测序仪均是基于链测序的原理,尚处于用户试用阶段,概念炒得非常热,得出的数据并不是十分理想,还有待进一步优化和改进。测序原理见下图。
参考文献:Clarke J, et al. Continuous base identification for single-molecule nanopore DNA sequencing. Nature Nanotechnology 4: 265-270 (2009).
五、其它技术


利用电子显微镜对DNA分子直接进行测序的设想在上世纪六七十年代已经出现并经过实验验证,其主要原理是利用重原子如汞、锇、金等对单核苷酸进行标记,PCR得到带标记的DNA双链,将之固定在基质上并拉伸成直线,用透射电镜 (Transmission electron microscope, TEM) 观察,从而利用重原子标记识别DNA上的碱基排列。TEM测序的优势在于长片段读取 (10-20Kb),且没有光学信号错误,但重原子标记是其目前亟需解决的问题,PCR过程可能引入错误的标记信号,同时TEM的使用和维护成本较高也是制约该技术的主要因素。ZS Genetic (MA, USA) 与哈佛大学合作,正在进行这一技术的开发,希望能将其用于病毒突变的快速检测、单体型测序和癌症研究等方面。
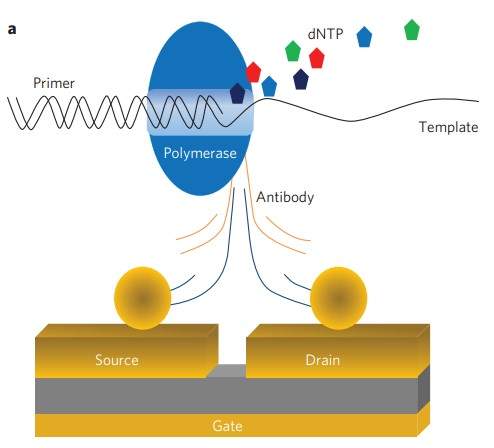
去年,台湾国立交通大学的Yu-Shiun Chen等利用DNA聚合酶、抗体和纳米金颗粒制备蛋白质晶体管 (protein transistor),并用于DNA测序的研究。这一方法思路新颖,研究成果发表在Nature Nanotechnology上之后立刻引起了强烈的反响,and争议。很多同行认为该方法有很多缺陷,以至于怀疑其实验结果的真实性。文章到目前为止仍然under investigation。但无论如何,这一成果仍为单分子测序提供了一种可能的方法。
研究人员首先在硅基质上制备了一对宽50nm,间距10nm的金电极,在原子力显微镜 (atomic force microscope, AFM)下将两个直径5nm的金颗粒分别放到两个电极的边缘,最后采用微流控的方法将抗体和DNA聚合酶自组装到预定位置,组成单分子蛋白质晶体管。通过检测DNA聚合酶合成DNA时电导率的变化,识别出DNA序列。据报道,利用该结构的蛋白质晶体管,能够检测到DNA聚合酶在合成DNA时由于电导率变化引起的电流改变,变化幅度约为3pA,且不同dNTP合成时会产生不同的电流特征,因此可以利用这一现象区分DNA的碱基排列。该方法的测序速率约为22nt每秒,单碱基准确度达到99.99%以上。
参考文献:
Bell DC, et al. DNA Base Identification by Electron Microscopy. Microsocpy and Microanalysis 18: 1049-1053 (2012).
Chen YS, et al. DNA sequencing using electrical conductance measurements of a DNA polymerase. Nature Nanotechnology 8: 452-458 (2013),
六、个人观点
目前来看,单分子测序技术还不够成熟,但潜力无限。Helicos倒闭,一是仪器性能平平,读长35bp,仅与Illumina早期的GA相当,测序质量也不算高,二来价格太贵,一台仪器100多万刀,比Hiseq2000还贵,真不是一般人用得起。PacBio市场占有率低,但凭借着无人可比的8.5Kb读长,在复杂基因组de nove、宏基因组研究等领域牢牢占据了一席之地。Oxford Nanopore性能有待提高,可是炒作很成功,吸引了众多眼球,包括IBM在内的多家公司都对它表现出极大的兴趣和期望,学术界也普遍认为纳米孔是最有希望的单分子测序技术。至于剩下的,都在实验室里折腾,原型机都还没做出来。
二代测序方兴未艾,三代测序已然来势汹汹。全球的科研、产业乃至资本力量都将目光投向了人类基因组的深入探索中。个体化医疗、肿瘤研究、遗传病、天赋基因,各种热门词汇不绝于耳,而这一切,都离不开最上游的测序技术。欧美发达国家又一次走在了最前沿,国内99.9%的测序数据都是由进口仪器所产出,包括华大在内的所有测序服务提供商均受制于illumina、Life(已被ThermoFisher收购)等测序巨头。三代测序,也许能够为我们基因组产业链的上游带来少许希望,台湾的研究成果就是一个很好的例证。另一方面,测序技术衍生的一系列技术和方法也未尝不是一个好的突破点,例如北京大学谢晓亮教授的MALBAC单细胞DNA扩增技术(虽然是在Havard搞出来的),已经是国际上应用最广的单细胞扩增技术之一。高大上的测序仪做不了,做一些小而精的试剂盒也前景光明。
OK~写完!小虫才疏学浅,难免有疏漏之处,还望各位虫友轻拍......欢迎查漏补缺,批评指正!
顶一下,感谢分享!
顶一下,感谢分享!