两个结构类似的分子做柔性势能面扫描,结果差别很大,可否帮忙看下怎么回事?
两个类似的分子,固定开环两个碳原子之间的距离,用关键词opt=modredundant,做开环反应的柔性势能面扫描,结果差别很大,可否帮忙看下怎么回事?
第一个分子能够从闭合状态顺利地优化到开环状态,高斯view打开看中间结构,中间文件过渡态一切正常,如下:
势能面扫描结果
https://muchongimg.xmcimg.com/os ... _1604036712_548.png
起始闭环结构
https://muchongimg.xmcimg.com/os ... _1604036788_386.png
曲线最高点-过渡态结构
https://muchongimg.xmcimg.com/os ... _1604036826_228.png
最终闭环结构
https://muchongimg.xmcimg.com/os ... _1604036854_507.png
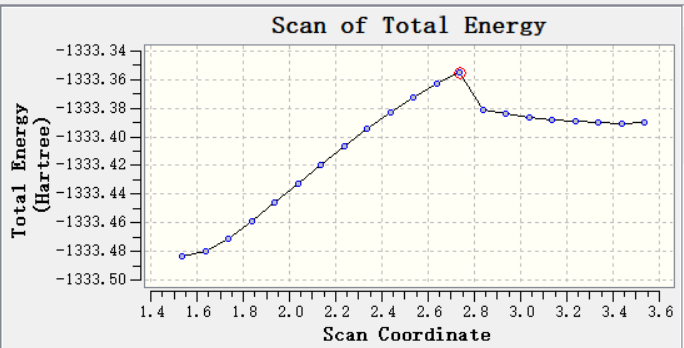
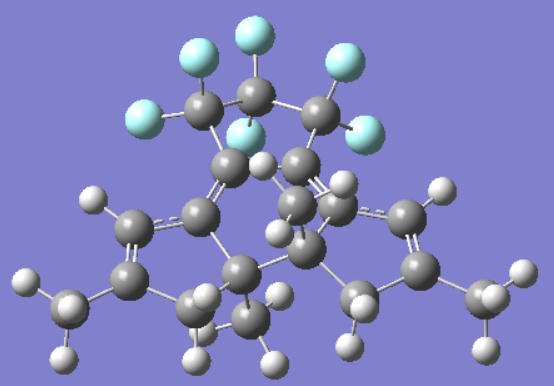
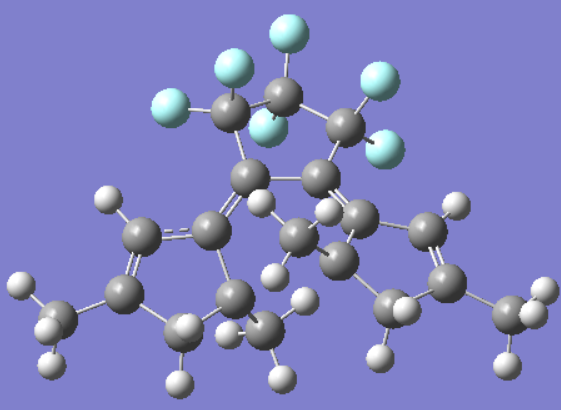
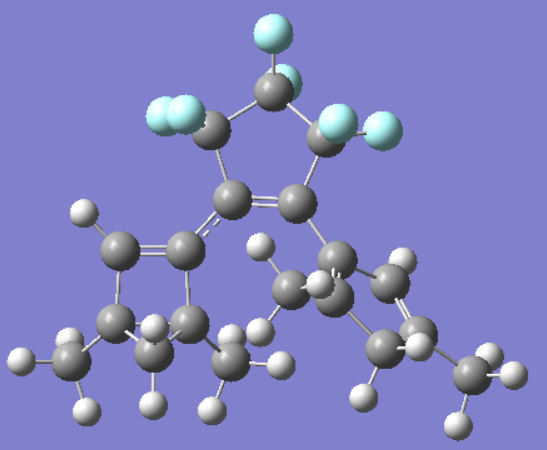
第二个分子能够从闭合状态顺利地优化到过渡态,从过渡态最高点之后,所有结构均发生扭曲变形,导致扫描不能进行到开环状态,详细结果如下:
势能面扫描结果
https://muchongimg.xmcimg.com/os ... _1604037000_277.png
起始闭环结构
https://muchongimg.xmcimg.com/os ... _1604037084_623.png
曲线最高点--过渡态正常结构
https://muchongimg.xmcimg.com/os ... _1604037128_409.png
过渡态之后--扭曲变形结构
这是第二个分子热不稳定吗?还是计算方法出了问题?
两个分子的开环状态和闭环状态都是稳定的,我已经得到稳定结构构型,为啥势能面扫描会出这样的问题呢?
接下来我该如何寻找分子2的过渡态呢?opt=ts已经试过了,不太行,不得已做的势能面扫描,我该如何改进势能面扫描呢?
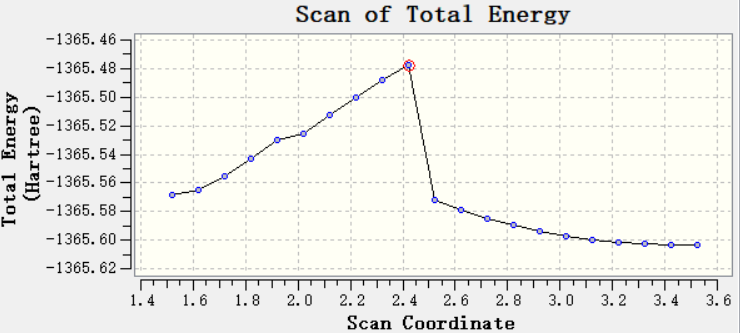
势能面扫描曲线.png
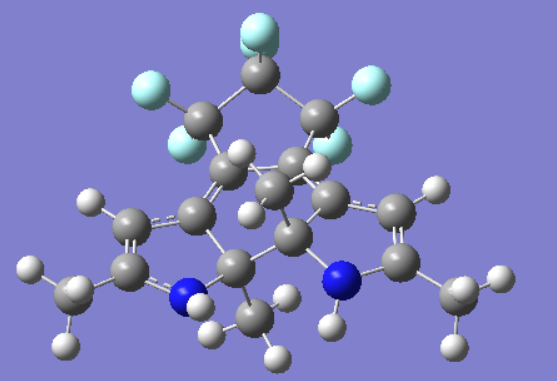
分子1起始闭环结构.png
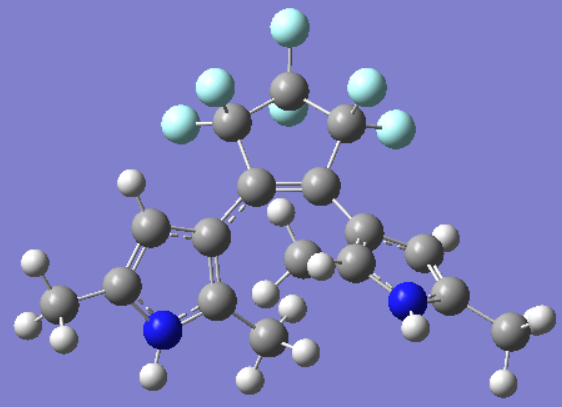
分子1中间过渡态结构.png
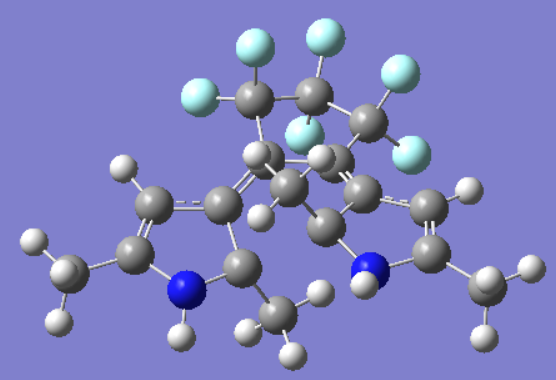
分子1结束闭环结构.png
分子2势能面扫描曲线.png
分子2起始闭环结构.png
分子2中间曲线最高点过渡态结构.png
分子2过渡态之后的扭曲变形结构.png![]() 返回小木虫查看更多
返回小木虫查看更多



 京公网安备 11010802022153号
京公网安备 11010802022153号
这个扫描是沿着两个原子间距离还进行的。而开环 往往不单纯是就是严格沿着两个原子之间的距离来进行开环的。单一一个内坐标变量的扫描不能完全重现开环过程。但是既然扫描已经出现顶点,是潜在的可能的过渡态结构。不妨就把两个过渡态都优化出来, 跑IRC曲线。如果过渡态结构优化正确。IRC曲线应该就能更好的体现开环的过程了。
扫描是沿着两个C-C原子间距离进行的
现在还有个问题就是opt=TS,优化不出来过渡态,
附件里分别是opt=TS优化的分子1和分子2的log文件,目前为止优化好多天了,一直没有结果
,
附件文件刚上传失败了,再传一次
那看起来顶点的地方不是 过渡态的那种顶点,而是结构有很大变化时候出现能能量突跃。