| 查看: 1462 | 回复: 5 | ||
| 当前只显示满足指定条件的回帖,点击这里查看本话题的所有回帖 | ||
[交流]
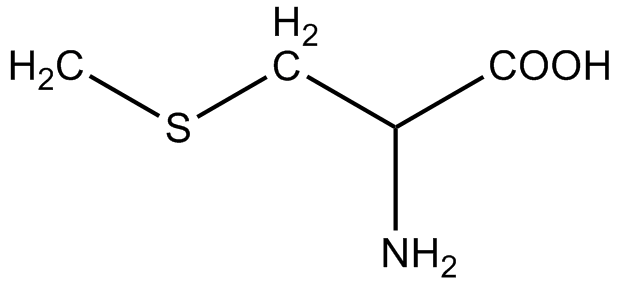
【求助】请教如何生成非天然序列的pdb用于zdock 已有3人参与
|
||
» 本主题相关价值贴推荐,对您同样有帮助:
 请教Elsevier期刊但非SCI的问题
已经有4人回复
请教Elsevier期刊但非SCI的问题
已经有4人回复
 【原创非首发】天使在哪里
已经有19人回复
【原创非首发】天使在哪里
已经有19人回复
 PVA用作陶瓷造粒时的粘结剂的制备
已经有33人回复
PVA用作陶瓷造粒时的粘结剂的制备
已经有33人回复
 关于鼓励应助的奖励办法征集意见
已经有13人回复
关于鼓励应助的奖励办法征集意见
已经有13人回复
 堇青石的生成温度
已经有7人回复
堇青石的生成温度
已经有7人回复
 请教各位如何降低混凝土中的气孔含量
已经有11人回复
请教各位如何降低混凝土中的气孔含量
已经有11人回复
 金属表面陶瓷涂层起泡是什么原因?
已经有31人回复
金属表面陶瓷涂层起泡是什么原因?
已经有31人回复
 【求助】BaTiO3,K0.5Na0.5NbO3, (Bi0.5K0.5)TiO3三种材料的介电常数
已经有10人回复
【求助】BaTiO3,K0.5Na0.5NbO3, (Bi0.5K0.5)TiO3三种材料的介电常数
已经有10人回复
5楼2010-06-13 18:42:52
2楼2010-06-13 08:28:55
tjegg
铁杆木虫 (著名写手)
- 应助: 26 (小学生)
- 金币: 6869.3
- 红花: 16
- 帖子: 2615
- 在线: 448小时
- 虫号: 338983
- 注册: 2007-04-05
- 性别: GG
- 专业: 抗肿瘤药物药理

3楼2010-06-13 16:27:47
4楼2010-06-13 18:39:31















 回复此楼
回复此楼