| 查看: 1497 | 回复: 3 | ||
[求助]
求教Gromacs计算蛋白-蛋白相互作用的top文件建立方法
|
||
|
|
||
hclwonder新虫 (正式写手)
|
|
|
|
2楼2017-04-15 08:44:52
|
|
|
|
3楼2017-04-15 21:14:39
|
|
|
hclwonder新虫 (正式写手)
|
||
|
4楼2017-04-15 22:06:14
|
|
| 4 | 1/1 | 返回列表 |
相关版块跳转
我要订阅楼主 Jay_LIN 的主题更新
| 4 | 1/1 | 返回列表 |














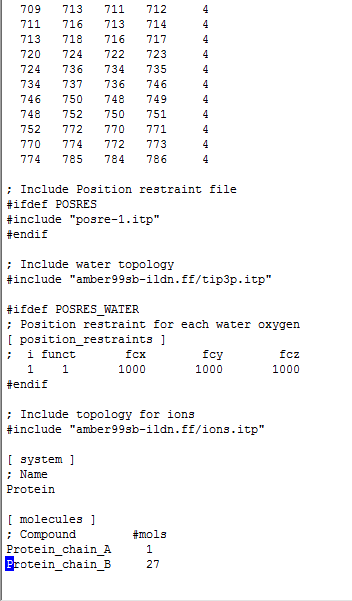
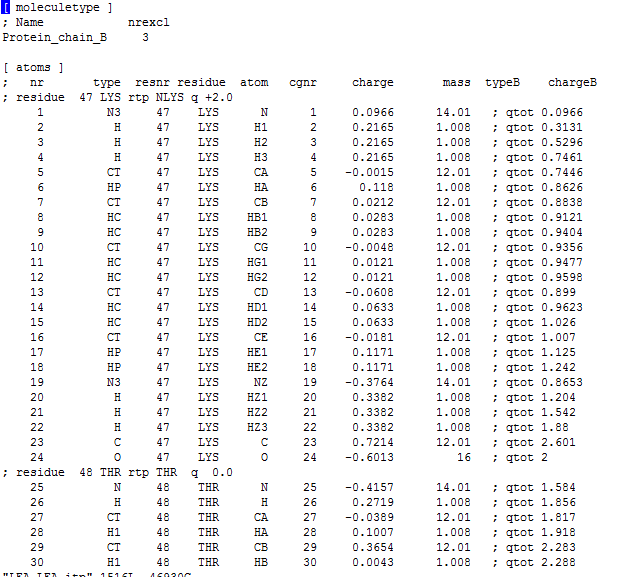
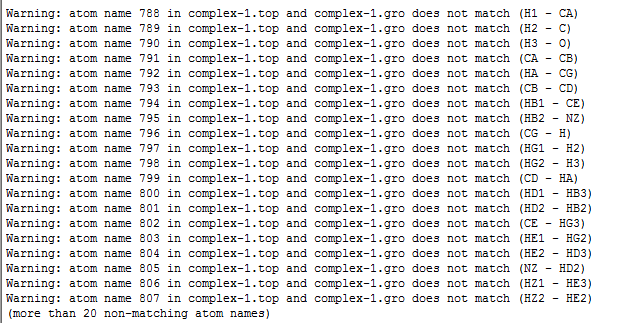
 。我最近在计算蛋白-蛋白相互作用时碰到了top文件的建立问题。图和一图二分别是我的蛋白-蛋白复合体系的top文件的头部与底部。我的想法就是将配体蛋白的itp(LEA-LEA.itp)include进top中。
。我最近在计算蛋白-蛋白相互作用时碰到了top文件的建立问题。图和一图二分别是我的蛋白-蛋白复合体系的top文件的头部与底部。我的想法就是将配体蛋白的itp(LEA-LEA.itp)include进top中。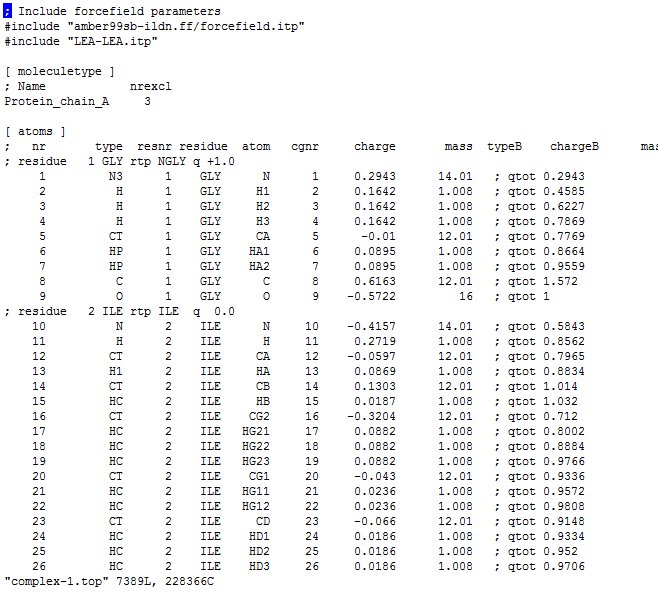
 谢谢大家了!)
谢谢大家了!) 回复此楼
回复此楼
