差异基因表达,GO分析,miRNA靶基因预测新思路
一、芯片差异基因分析
1. 芯片数据收集
在 NCBI GEO数据库下载 。
2、做差异分析
使用limma R包计算正常组织和病组织的差异表达情况

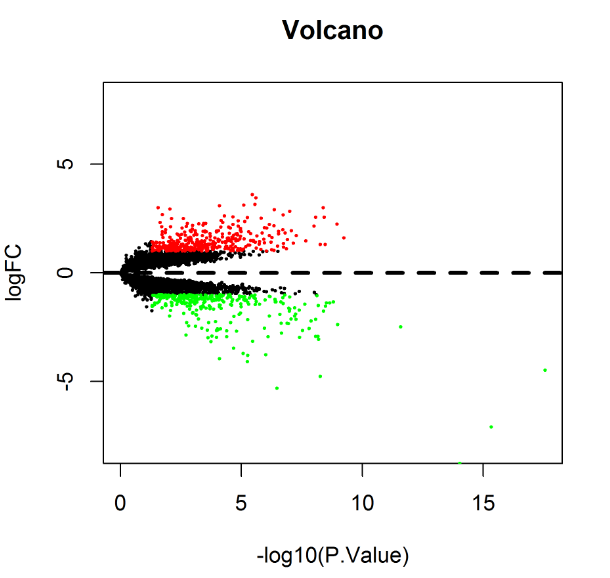
3、绘制火山图
4、绘制热图
使用pheatmap R包对差异基因进行聚类分析

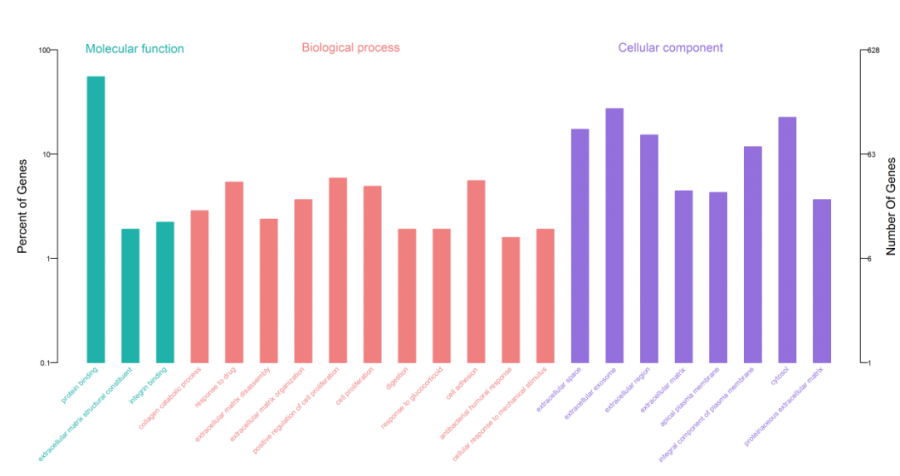
5、差异基因GO分析
使用DAVID对差异基因进行GO功能富集分析
6、KEGG通路富集分析
使用DAVID对差异基因进行KEGG功能富集分析

7、蛋白互作网络
使用 String在线工具对得到的差异表达基因进行蛋白质与蛋白质相互作用关系的构建
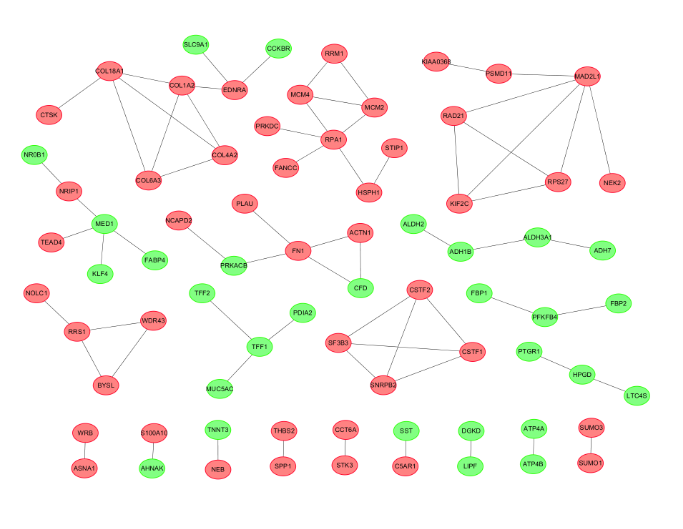
8、子网络
使用 cytoscape 软件的 MCODE[9]插件,获得网络的重要模块。
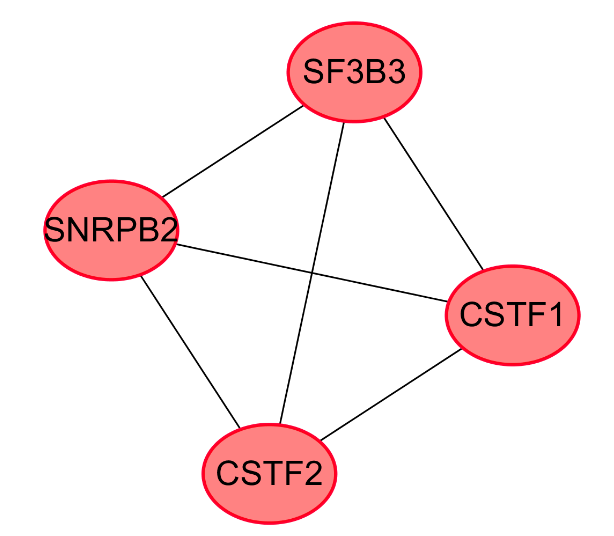
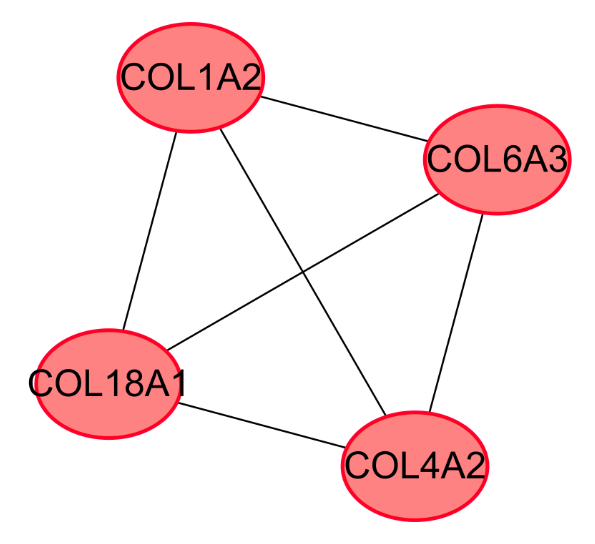

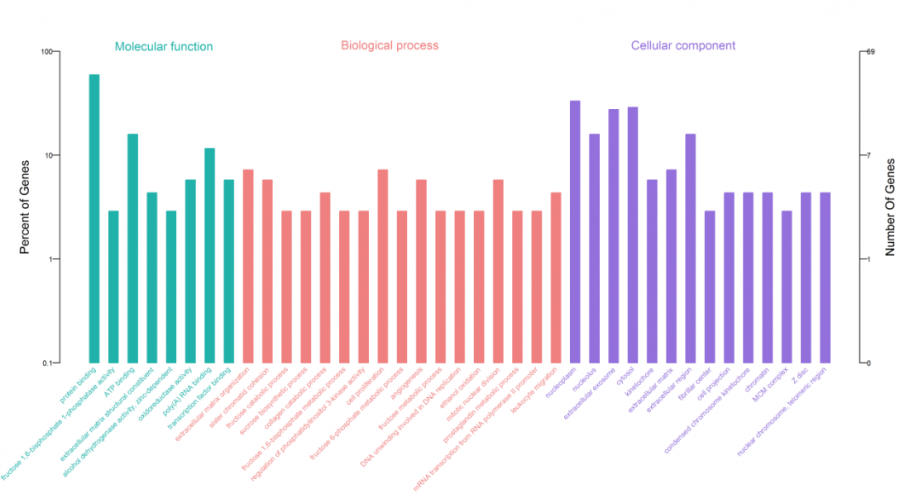
9、每个子网络的GO分析
使用DAVID对每个子网络差异基因进行GO功能富集分析

二、miRNA靶基因分析
1. 芯片数据收集
在 NCBI GEO数据库下载 。
2. 数据预处理及差异分析
使用limma R包计算差异miRNA情况
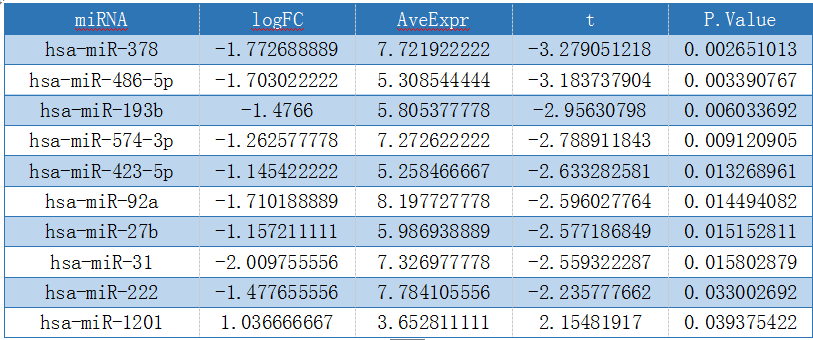
3、火山图
4、绘制热图
使用pheatmap R包对差异miRNA进行聚类分析。
5、靶基因预测
对差异表达 miRNA 进行靶基因预测分析。

6、miRNA-gene调控网络
使用 cytoscape 软件进行miRNA-gene 调控网络图构建。
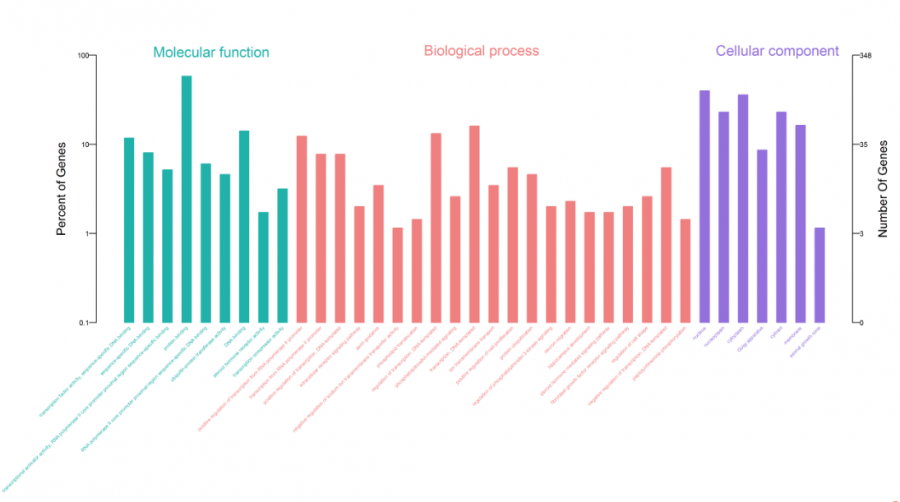
7、miRNA靶基因GO功能分析
使用DAVID对差异miRNA靶基因基因进行GO功能富集分析
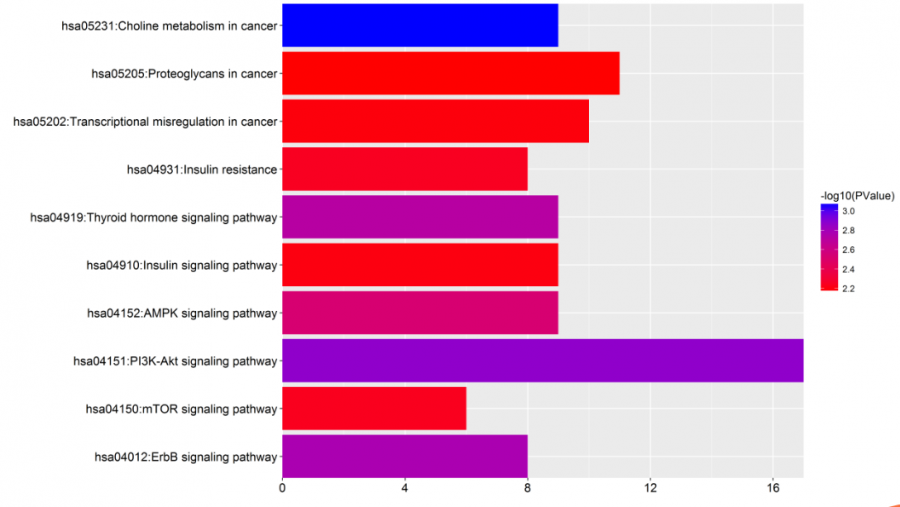
8、miRNA靶基因KEGG通路富集分析
使用DAVID对差异miRNA靶基因基因进行GO功能富集分析
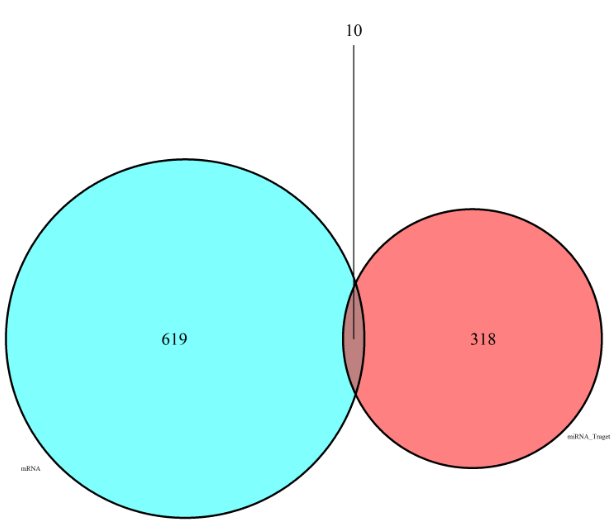
9、差异基因与miRNA靶基因做交集
差异基因与miRNA靶基因做交集,得到基因芯片和miRNA芯片预测的靶基因的交集基因


本报告为原创,欢迎交流指正,需要引用或转载文章的内容或图片,请联系作者,谢谢!
一、下载数据
1、网页进入GEO(https://www.ncbi.nlm.nih.gov/geo/)
选择Search for Studies at GEO DataSets
2、进入芯片搜索框,输入需要搜索的关键词,比如做胃癌的,输入gastric cancer
3、结果页面找到和研究相关的芯片数据
4、进入芯片数据页面,下载DataSet full SOFT file
5、阅读芯片相关介绍,需要重点关注实验内容,包括实验组和对照找,实验数目等




二、基因差异化表达
1、解压下载的GDS1210_full.soft.gz压缩包,得到GDS1210_full.soft
2、用UeDit打开查看GDS1210_full.soft文件
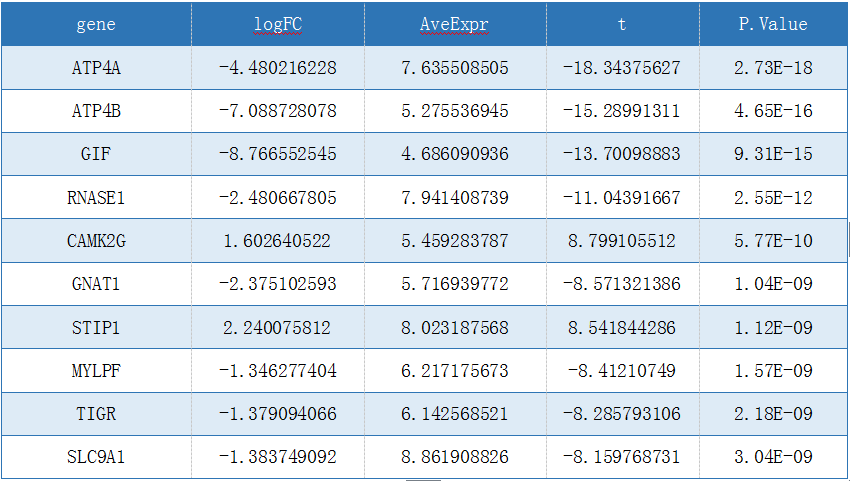
3、使用 R 软件的 limma 包计算,选择阈值为|logFC|>1 且 P<0.05。(R包可找ID的Q)
4、使用limma R包计算差异表达情况(|logFC|>1 & P<0.05),共找到628个满足条件的差异基因。其中350个基因上调,278个基因下调。
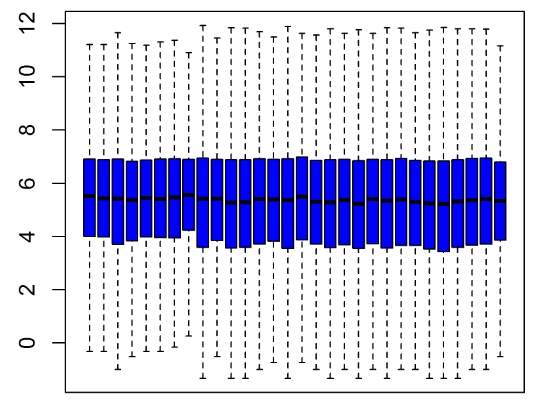
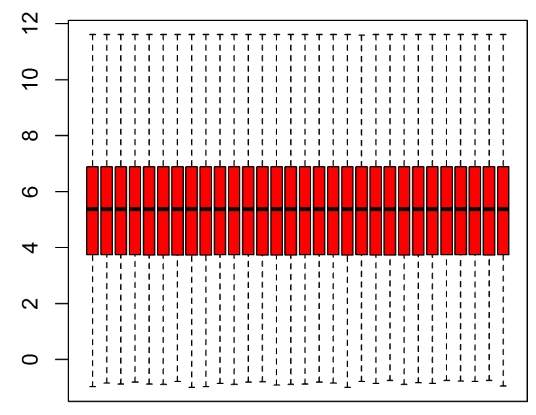
归一化前后对比


18.png
21.png
[ Last edited by QQ1556760763 on 2017-3-12 at 21:35 ]![]() 返回小木虫查看更多
返回小木虫查看更多



 京公网安备 11010802022153号
京公网安备 11010802022153号
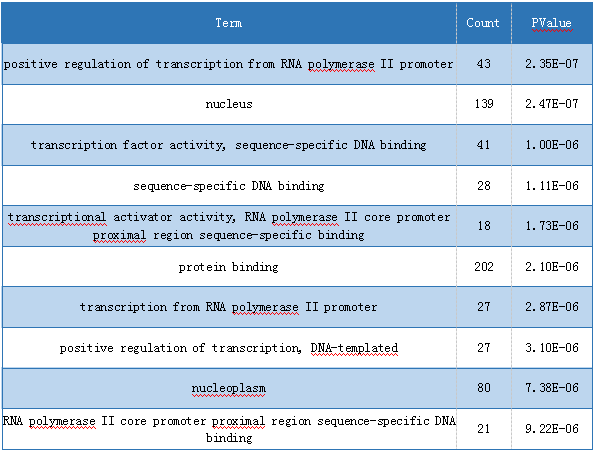
GO功能分析



 ,
,
一、DAVID在线分析(PValue< 0.05被作为筛选条件。)
1、进入DAVID(https://david.ncifcrf.gov/)
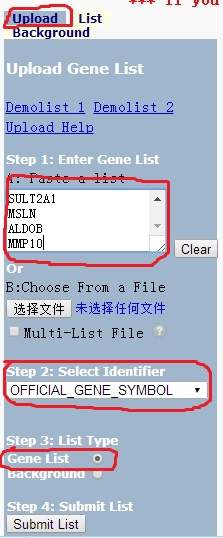
2、Upland-Faste a list输入需要做GO分析的差异基因
3、List选择物种
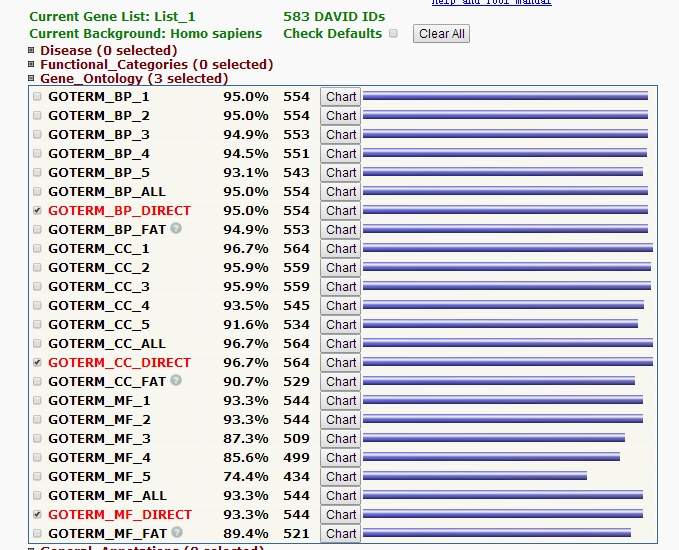
4、在线分析得到GO数据
5、下载数据
二、得到389个相关的GO,使用R脚本生成差异基因GO富集结果
请问如何使用R脚本生成差异基因GO富集结果? 谢谢楼主
我今天刚开始认真学习生物信息学,希望多多指教,如何学好生物信息学,大恩不言谢(;`O?)o
GO功能富集的R脚本是某宝店家给的,人家说有版权的
可以的,交流学习可以加小木虫的账号Q
我刚做了两个菌的转录组学分析,然后用嘴低端的方法自己一个一个去分类,突然看到这个帖子,楼主可否提供一些更为详细的资料,处理一下转录组数据,看这个感觉有点看不明白啊
新手,基因芯片数据如何预处理?